- “La prescripción de ejercicio físico debe ser una prioridad de salud pública” tanto para prevenir como para tratar múltiples enfermedades, según el informe dirigido por Mikel Izquierdo, catedrático de la UPNA e investigador en Navarrabiomed
La Universidad Pública de Navarra (UPNA) ha liderado un consenso global sobre ejercicio físico, un acuerdo basado en la evidencia científica y firmado por múltiples especialistas internacionales en esta área. El panel de 34 investigadores de veintiséis instituciones dirigido por Mikel Izquierdo Redín, catedrático de la UPNA, parte del hecho de que la esperanza de vida ha aumentado considerablemente y el desafío no es solo vivir más años, sino hacerlo con buena salud y autonomía. En este contexto, la actividad física desempeña un papel crucial. “El ejercicio es la intervención más efectiva, segura y accesible para reducir el impacto de las enfermedades relacionadas con la edad", destaca el documento. Sus autores añaden que “el ejercicio debe ser una prioridad de salud pública”, no solo como medicina preventiva, sino también como tratamiento para múltiples enfermedades.
Acceso libre al consenso global sobre ejercicio
El “Consenso global sobre recomendaciones óptimas de ejercicio para promover una longevidad saludable en personas mayores” se ha publicado en la revista científica “The Journal of Nutrition, Health and Aging” y está disponible para consultar “online” en acceso libre.
El documento ofrece respuestas claras y basadas en la evidencia. “El ejercicio físico no solo mejora la calidad de vida de los adultos mayores, sino que también es una herramienta esencial para prevenir enfermedades y fomentar una longevidad saludable”, expone el informe, que hace un llamamiento a gobiernos, profesionales sanitarios, educadores físico-deportivos, entrenadores y a la sociedad en general para adoptar este enfoque como una estrategia efectiva de promoción de la salud y bienestar.
“Estas recomendaciones, respaldadas por un panel internacional de expertos, establecen un marco clave para transformar el concepto de envejecimiento. Además, promueven intervenciones específicas y basadas en la evidencia que puedan aplicarse en la práctica clínica diaria, contribuyendo así al bienestar integral de una población cada vez más longeva”, afirma Mikel Izquierdo, investigador principal al frente de especialistas en envejecimiento, salud y ejercicio físico de dieciséis países. Entre ellos, figuran también los investigadores de la UPNA Antonio García Hermoso, Robinson Ramírez Vélez y Mikel López Sáez de Asteasu.
Vivir más años con buena salud
En un contexto donde la esperanza de vida sigue creciendo, el reto no es solo vivir más años, sino hacerlo con calidad de vida. Según el panel internacional autor del consenso, la actividad física desempeña un papel crucial en este proceso. “El ejercicio es la intervención más efectiva, segura y accesible para reducir el impacto de las enfermedades relacionadas con la edad”, expone el documento.
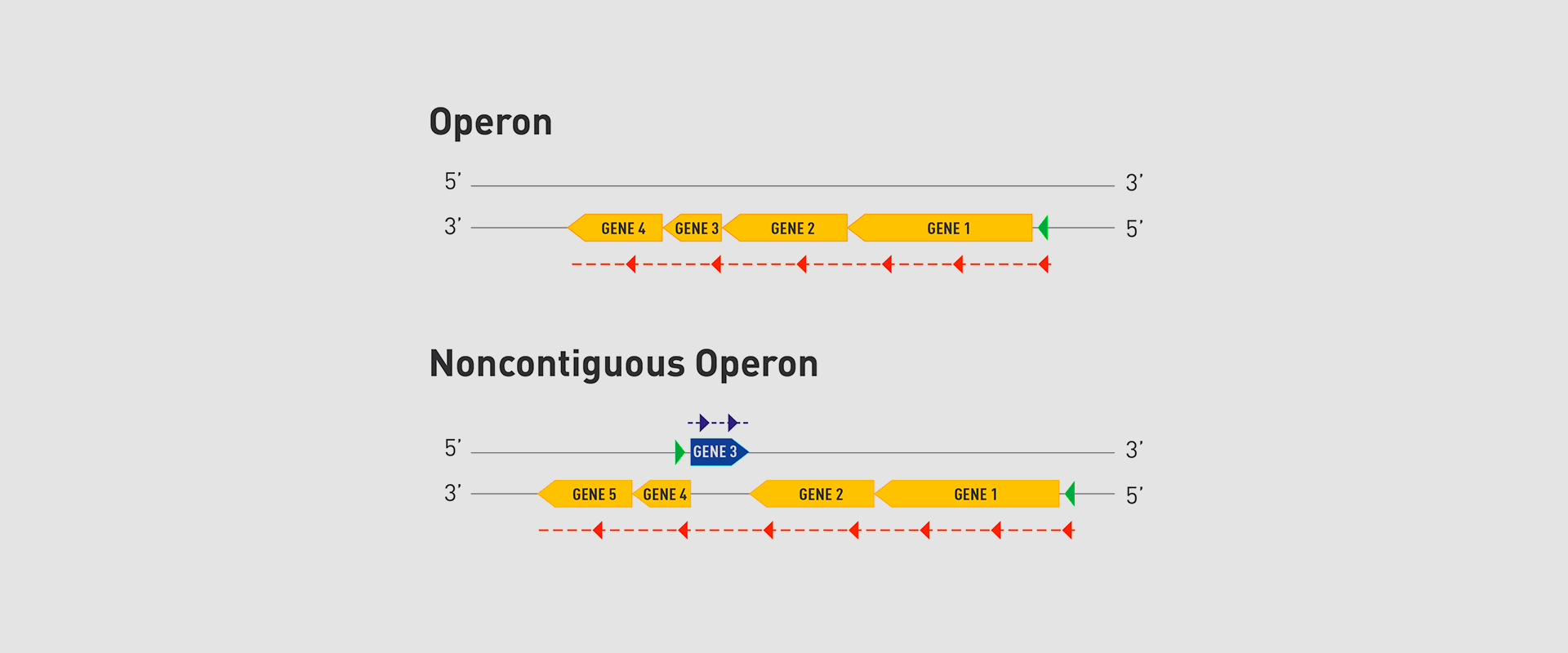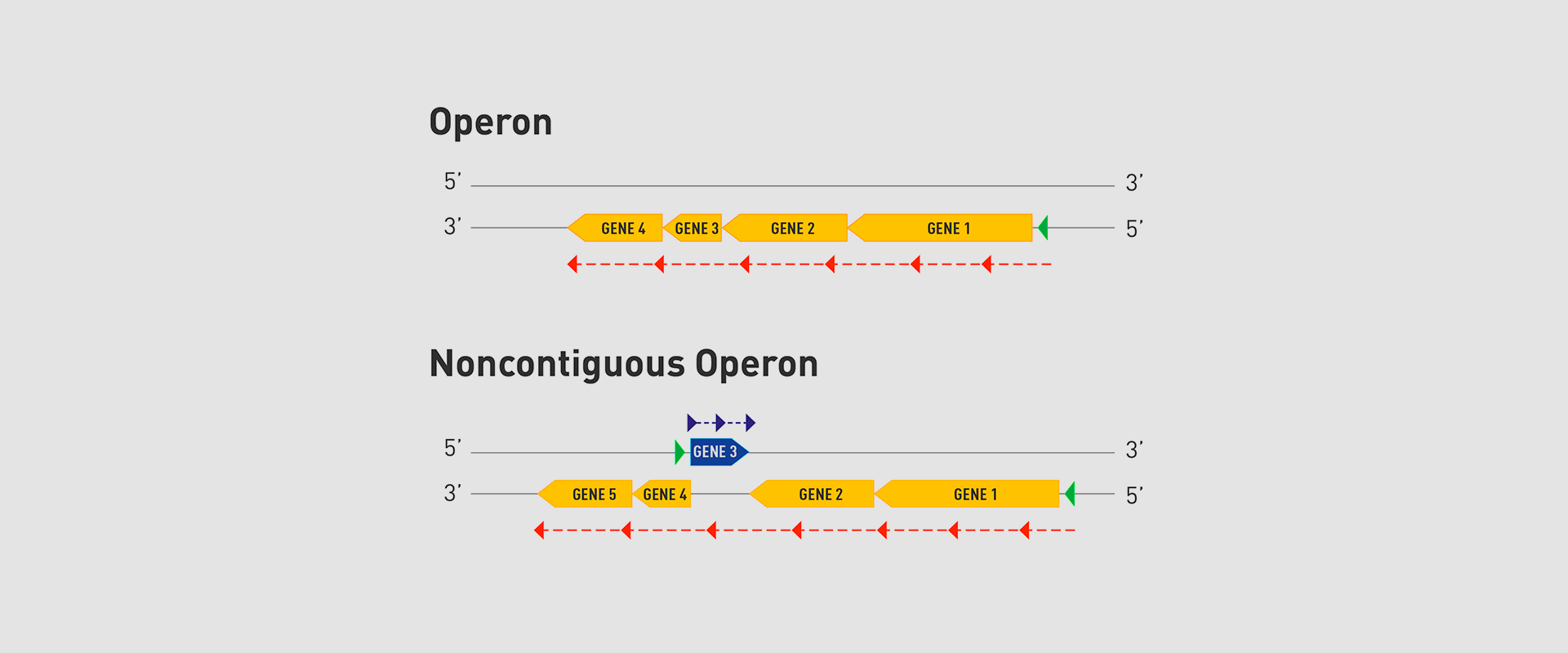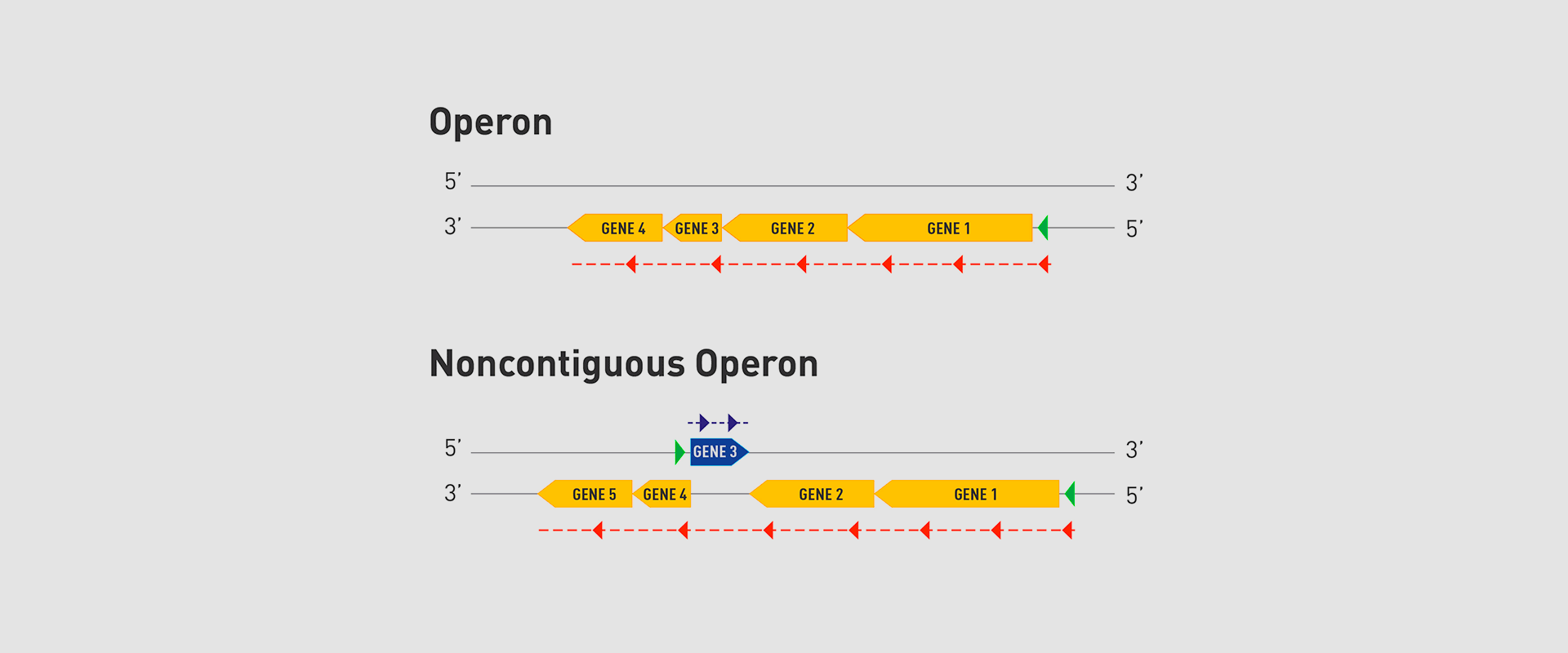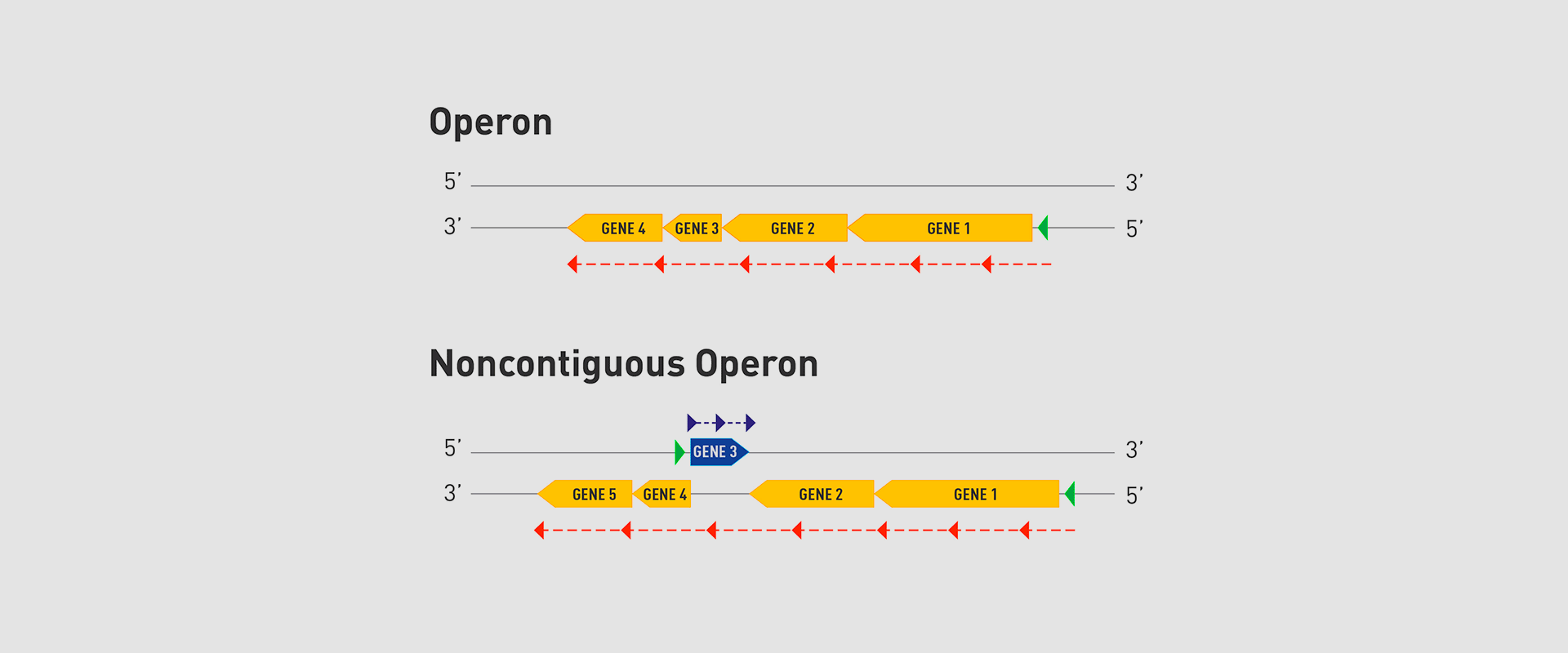
Uno de los puntos más innovadores es el diseño de una secuencia estructurada para la prescripción de ejercicio en adultos mayores. Este proceso comienza con una evaluación inicial integral para identificar las necesidades y capacidades específicas de cada persona, seguida de un plan detallado que incluye la secuenciación, progresión, especificidad y personalización, según las preferencias del paciente, con el fin de mejorar la adherencia.
El documento también subraya la importancia de respetar las preferencias individuales y diseñar planes personalizados que impulsen un mayor compromiso. Asimismo, se incorporan estrategias de apoyo cognitivo y social para reforzar la adherencia a largo plazo.
Otro aspecto destacado es el énfasis en el entrenamiento funcional, que prioriza ejercicios de equilibrio y prevención de caídas para garantizar la seguridad, mejorar la movilidad y reducir los factores de riesgo asociados al envejecimiento.
Recetas individualizadas para un envejecimiento saludable
Uno de los aspectos más relevantes del consenso es su enfoque personalizado. Las recomendaciones generales sobre ejercicio físico se adaptan a las necesidades y características individuales de cada persona, considerando su estado de salud, nivel de actividad física y metas personales. “No existe una receta única para todos. Diseñar programas de ejercicio individualizados es esencial para maximizar los beneficios y garantizar tanto la seguridad como la eficacia en las personas mayores”, detalla el documento.
Otra innovación significativa es la inclusión de programas de ejercicio multicomponente que integran tareas cognitivas para fortalecer la resiliencia física y mental. Este enfoque integral busca mitigar la fragilidad y mejorar la autonomía. Entre las recomendaciones más relevantes, sobresalen las directrices para implementar entrenamientos de fuerza muscular progresivos de alta intensidad, entrenamiento de potencia y ejercicios por intervalos de alta intensidad, que han demostrado ser prometedores en el mantenimiento de la salud cardiovascular y muscular en adultos mayores.
Asimismo, el documento resalta que el impacto del ejercicio trasciende la salud individual. Promover el ejercicio físico regular no solo beneficia al individuo, sino que también “fortalece a las comunidades al reducir la dependencia y mejorar el sentido de pertenencia y la participación social”, según recoge el consenso global.
El ejercicio: medicina preventiva y terapéutica
El ejercicio basado en evidencia científica representa una herramienta poderosa para promover un envejecimiento saludable, prevenir enfermedades relacionadas con la edad y tratar condiciones como la fragilidad y la sarcopenia, según el grupo de especialistas. Las implicaciones sociales van más allá de la reducción de costes sanitarios, pues contribuye a una mejor calidad de vida y a una mayor independencia funcional de las personas mayores, a juicio del panel internacional.
“Ha llegado el momento de integrar el ejercicio físico como un pilar central de la atención geriátrica y de los sistemas de salud, con el fin de extender la vida activa y el bienestar de las futuras generaciones —indica Mikel Izquierdo—. Este consenso aboga por programas de ejercicio personalizados y basados en evidencia, adaptados a las capacidades y necesidades de cada persona mayor, independientemente de su edad, condición física o nivel de fragilidad inicial”.
De hecho, la fragilidad no debe verse como “una contraindicación para el ejercicio, sino como una de las principales razones para prescribirlo”, concluye el informe.